Die Diskussion über multiresistente Krankenhauskeime hat es wieder gezeigt: Es fehlt derzeit noch an neuen Antibiotika-Wirkstoffen, die beispielsweise auch bei resistenten Bakterien wirken. Mit einem neu entwickelten Verfahren könnten solche neuartigen antimikrobiellen Wirkstoffe aufgespürt werden. Diese wirken gezielt bei der Ribosomen-Entstehung ein und gelten daher als ideale Kandidaten für Antibiotika.
Bakterielle Ribosomen sind Ribonukleoproteinpartikel, die im Wesentlichen aus 54 Proteinen und drei ribosomalen RNAs (rRNA) bestehen. Ribosomen spielen bei der Proteinbiosynthese, d.h. der Neubildung von Proteinen in Zellen, die zentrale Rolle. Der hierarchische und genau gesteuerte Entstehungsprozess von Ribosomen in der lebenden Zelle wird als Ribosomen-Assemblierung bezeichnet und ist vergleichsweise wenig erforscht. In den Augen vieler Experten stellen frühe Prozesse in der Ribosomen-Entstehung attraktive Ziele für antimikrobielle Wirkstoffe dar. Neben Biogenesefaktoren (nicht ribosomale Proteine, die bei der Ribosomen-Assemblierung mitwirken) sind auch ribosomale Proteine ein potenzielles Ziel. Wirkstoffkandidaten wären beispielsweise kleine Moleküle, welche die Aktivität von Biogenesefaktoren und ribosomalen Proteinen beeinflussen oder die Bildung oder Auflösung bestimmter Strukturen in der rRNA behindern.
Die rRNA gilt als besonders interessanter Angriffspunkt, weil sie in Bakterien von Genen codiert wird, die in mehrfacher Kopienzahl vorliegen. Das Risiko einer Resistenzbildung wäre somit stark vermindert.
Bislang gibt es aber keine Antibiotika auf dem Markt, die primär die Entstehung der Ribosomen hemmen. Die systematische Suche nach solchen Substanzen wird durch die Tatsache erschwert, dass bislang kein geeignetes Screening-Verfahren existierte.
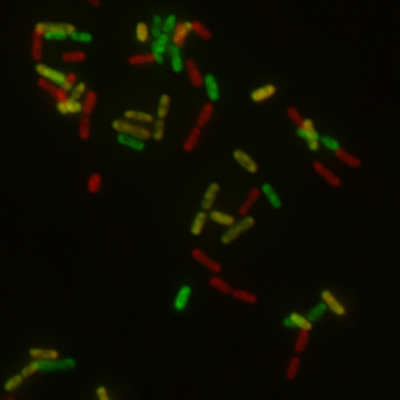
Wissenschaftlern der Universität Konstanz ist es kürzlich gelungen, Bakterienstämme mit Fluoreszenz-markierten, ribosomalen Untereinheiten herzustellen, die Wildtyp-ähnliche Wachstumseigenschaften zeigen und einen intakten Translationsapparat zu Synthese von Proteinen aufweisen.
Prof. Elke Deuerling vom Institut für Molekulare Mikrobiologie und Dr. Rainer Nikolay, der mittlerweile an der Berliner Charité forscht, entwickelten zunächst Bakterienstämme mit unterschiedlichen Fluoreszenz-Markierungen. Anschließend konnten sie mit dem neuen Screening-Verfahren aufzeigen, dass die Positionierung der Fluorophore es ermöglicht, Störungen bei der Ribosomen-Assemblierung durch ein Fluoreszenz-basiertes Ausleseverfahren in vivo zu ermitteln. Das Verfahren wurde für die Nutzung von Mikrotiter-Platten optimiert und eignet sich deshalb für die Durchführung von Hochdurchsatz-Screenings (HTS).
Mit diesem neu entwickelten Verfahren könnten neuartige Wirkstoffe identifiziert werden, die die Ribosomen-Entstehung hemmen. Außerdem könnten der Grundlagenforschung neue Werkzeuge zur Verfügung stehen und gleichzeitig die Basis für neuartige antimikrobielle Leitsubstanzen geschaffen werden.
Die Technologie-Lizenz-Büro (TLB) GmbH betreut die Patentierung der Erfindung und ist mit der Lizenzierung und Vermarktung der Innovation beauftragt.
Weitere Informationen erteilt TLB-Innovationsmanagerin Dr. Uta Weirich (uweirich(at)tlb.de).